引言
全基因组关联分析(Genome-Wide Association Study, GWAS)是遗传学领域研究复杂性状和疾病遗传基础的强大工具。传统GWAS主要依赖SNP芯片技术,而随着测序技术的飞速发展,全基因组测序(Whole-Genome Sequencing, WGS)逐渐成为GWAS研究的新选择。
WGS-GWAS相比传统GWAS到底有哪些优势呢?
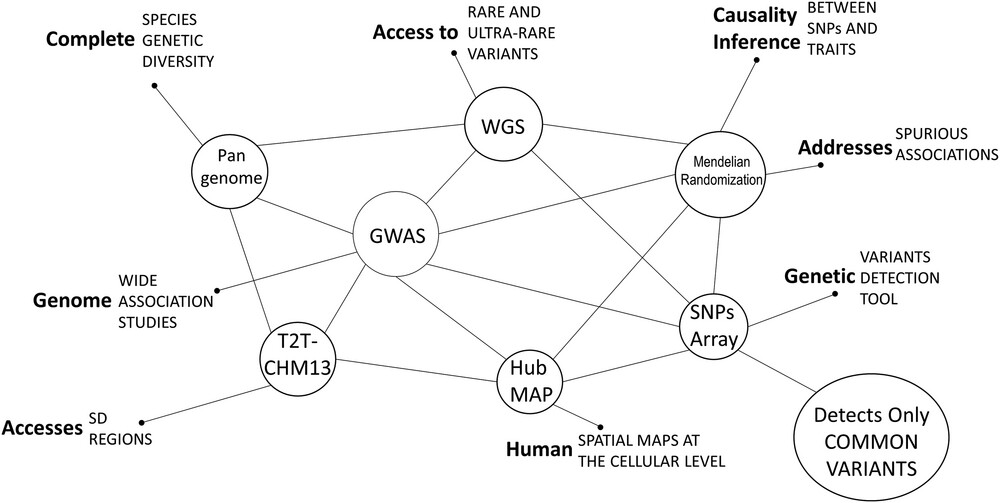
图片来自:Interconnected techniques in genome-wide association studies (GWAS) research
传统GWAS的局限性
传统GWAS使用SNP芯片技术,虽然在过去取得了巨大成功,但也存在一些明显的局限性:
变异覆盖有限:SNP芯片只能检测预先设计的常见变异位点(SNP),无法发现新的或罕见的遗传变异。
分辨率低:由于芯片探针数量有限,难以精确定位与疾病相关的具体变异。
罕见变异检测能力弱:主要针对常见变异(MAF > 0.05),对罕见变异(MAF < 0.01)的捕捉能力不足。
因果变异难识别:通常只能找到与疾病相关的标记SNP,而非真正的致病变异。
结构变异检测受限:SNP芯片专注于单核苷酸变异,对插入、删除等结构变异的检测能力较差。
群体特异性问题:芯片设计基于特定人群数据,可能不适用于其他群体。
数据质量依赖技术:依赖杂交技术,易受杂交误差影响。
灵活性低:分析局限于芯片预设的变异类型和范围。
这些局限性使得传统GWAS在研究复杂疾病的遗传机制时受到一定制约。
WGS-GWAS的优势
相比之下,使用全基因组测序数据进行GWAS(WGS-GWAS)带来了革命性的改进,以下是其主要优势:
1. 全面的遗传变异覆盖
WGS能够测定整个基因组的DNA序列,覆盖所有类型的遗传变异,包括SNP、罕见变异以及插入、删除等结构变异。
2. 更高的分辨率
WGS的分辨率可以精确到单个碱基对级别,帮助研究者更准确地定位与性状或疾病相关的遗传变异,避免传统GWAS中模糊定位的问题。
3. 罕见变异的检测能力
罕见变异(MAF < 0.01)在复杂疾病中可能扮演重要角色。WGS能够全面捕捉这些低频变异,为研究提供新的突破口。
4. 直接识别因果变异
传统GWAS通常找到的是与疾病关联的标记位点,而WGS可以直接检测潜在的因果变异,提升对疾病机制的理解。
5. 结构变异的捕捉
WGS不仅限于SNP,还能检测插入、删除、倒位等结构变异,这些变异可能对性状和疾病有重要影响。
6. 群体特异性变异的研究
WGS不受芯片设计的群体偏见限制,能发现特定人群特有的变异,为跨人群遗传研究提供支持。
7. 更高的数据质量
WGS通过直接测序DNA生成数据,相比SNP芯片的杂交技术,减少了技术误差,结果更可靠。
8. 分析灵活性
WGS数据包含整个基因组信息,研究者可以根据需要选择不同的变异类型和分析策略,灵活性远超传统GWAS。
Take-Home Message
总的来说,使用全基因组测序数据进行GWAS在变异覆盖、分辨率、罕见变异检测、因果变异识别、结构变异分析、群体特异性、数据质量和灵活性等方面都显著优于传统GWAS。这些优势使得WGS-GWAS在探索复杂性状和疾病遗传基础时展现出更大的潜力,为精准医学和遗传学研究开辟了新的可能性。